
Windows实用设置工具 v3.6.5 中文绿色单文件版
- 大小:80KB
- 分类:系统辅助
- 环境:Windows
- 更新:2025-01-16
热门排行
简介
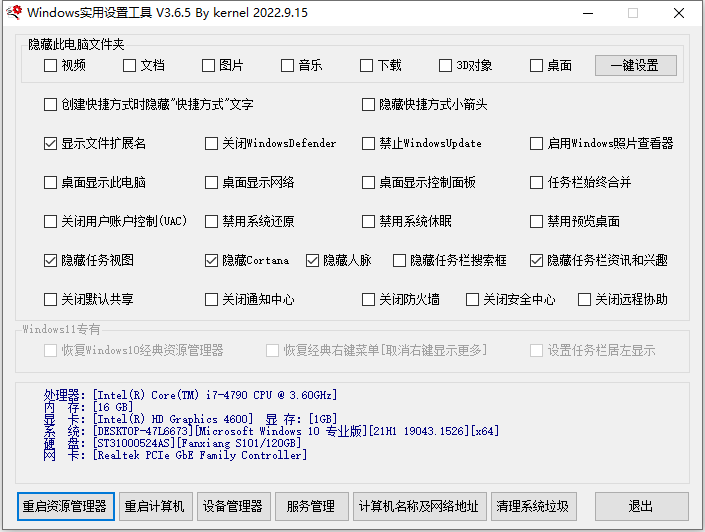
Windows实用设置工具是一款功能全面的系统设置辅助软件,适用于Windows 10、Windows 11等多个版本。它可以帮助用户快速进行系统设置,提升电脑使用效率。软件提供快速访问此电脑、控制面板、网络等选项的便利。启用或禁用某些默认系统服务,如关闭系统还原、休眠等。还有硬件信息显示,显示显卡、CPU、主板等硬件信息,方便用户了解系统情况。

系统要求
操作系统:Windows 10/11
功能亮点
1、定制开始菜单:可以添加或删除开始菜单选项, Keep实用设置工具提供了多种选择。
2、自定义任务栏:可以设置显示或隐藏任务栏图标,调整合并任务栏按钮等,满足个性化需求。
3、快速访问设置:提供快速访问此电脑、控制面板、网络等选项,方便设置。
4、文件选项设置:可以显示或隐藏文件扩展名、文件夹选项等,方便用户识别文件。
5、系统服务设置:可以启用或禁用某些默认系统服务,如关闭系统还原、休眠等。
6、界面显示设置:可以隐藏或关闭任务视图、搜索框、Cortana等界面元素。
7、安全和网络设置:可以关闭防火墙、用户账户控制等,增强系统安全性。
8、硬件信息显示:可以显示显卡、CPU、主板等硬件信息,了解系统情况。
软件功能
界面设计
简洁化的界面设计和完美的理念,连最小化都取消。打开界面只有4个按钮,让软件更易用,操作足够简单。并且它拥有众多皮肤,在简洁的界面上释放最大的功能。
理念
既然优化软件是为了加快电脑速度,为什么大部分优化软件关闭后在后台托盘,拖慢电脑呢?天威优化助手T7关闭后直接退出,不进入后台托盘。为你节省更多的内存。同时你可以自己为它随意开发你需要的应用或使用其他用户共享的应用。
软件提供了系统优化和清理、进程管理、木马查杀、硬件检测,管理中心,五大功能模块及数个附加的工具软件。
软件特色
优化电脑硬件,提升下载速度;
支持几乎所有网络连接类型如电缆、DSL、u - verse,Fios,拨号连接等;
减少程序的加载时间,大大提升电脑反应速度;
软件亮点
常见电脑问题一键修复,修完见效;
一次修复,长期不复发,坚持不定期修复,终身享受高效运行速度;
内置自动更新功能,无需打开网站更新;
更新日志
v3.6.5
1、软硬件信息显示加入:显卡名称及显存大小显示。
2022.8.25更新3.6.2版本:
加入Windows11的3个设置选项:
一、恢复windows10的经典资源管理器界面。
二、恢复经典的右键菜单。
三、设置任务栏居左或者居中
四、加入打开设备管理器,打开服务管理快捷按钮。
五、重启计算机加入提示,防止误点直接重启了。













 chrome下载
chrome下载 知乎下载
知乎下载 Kimi下载
Kimi下载 微信下载
微信下载 天猫下载
天猫下载 百度地图下载
百度地图下载 携程下载
携程下载 QQ音乐下载
QQ音乐下载